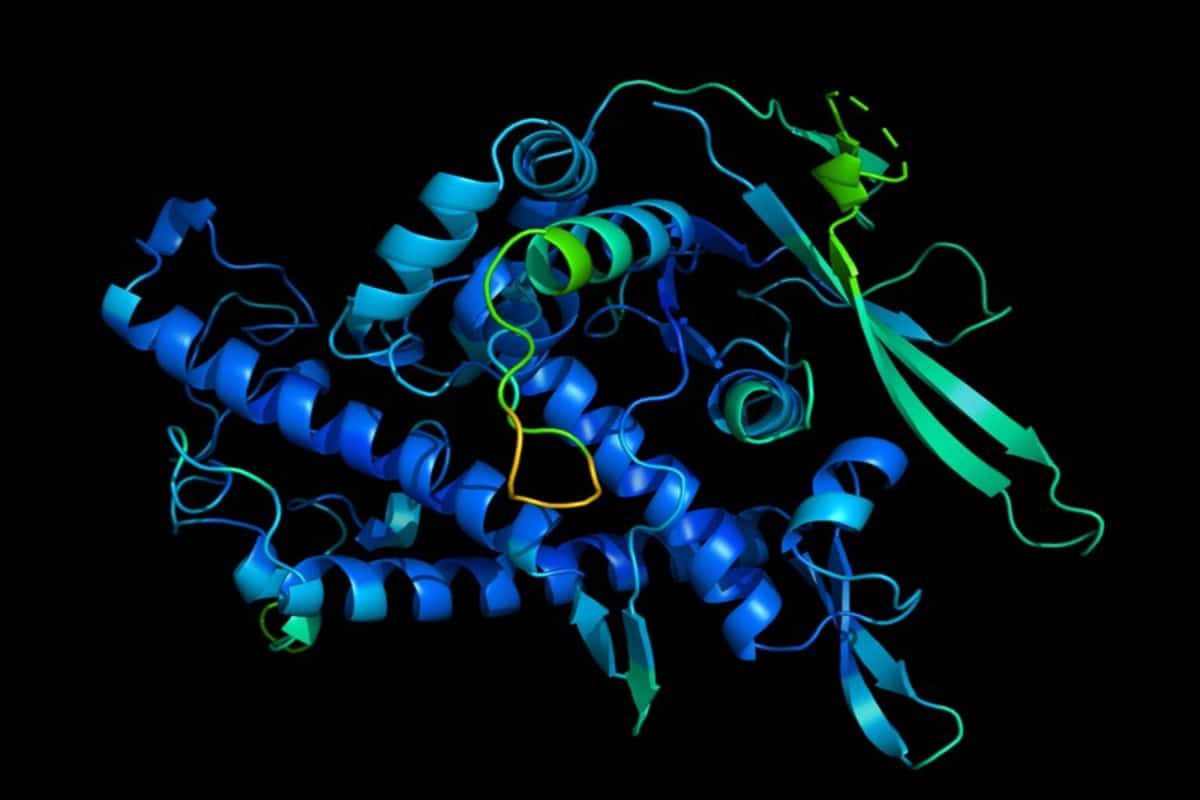
Una inteligencia artificial de la compañía de Google, DeepMind, logró resolver un enigma para la biología al determinar la forma en tercera dimensión (3D) de una proteína a partir de su secuencia de aminoácidos.
El programa que llevó a cabo la resolución del desafío se llama AlphaFold, y superó a 100 equipos en un desafío de predicción de la estructura de proteínas denominado Evaluación Crítica de la Predicción de Estructuras (CASP por sus siglas en inglés).
John Moult, biólogo computacional de la Universidad de Maryland, en College Park y cofundador de CASP, dijo para la revista científica Nature que "este era un gran problema... (y) en cierto sentido, el problema está resuelto".
Según la página oficial de DeepMind, estos resultados abren el potencial para que los biólogos utilicen la predicción de estructuras computacionales como una herramienta central en la investigación científica.
"Nuestros métodos pueden resultar especialmente útiles para clases importantes de proteínas, como las proteínas de membrana , que son muy difíciles de cristalizar y, por lo tanto, difíciles de determinar experimentalmente", se lee en el comunicado.
¿Qué son las proteínas?
La capacidad de predecir con precisión las estructuras de las proteínas a partir de su secuencia de aminoácidos sería de gran ayuda para las ciencias naturales y la medicina, ya que las proteínas son esenciales para la vida, son moléculas grandes y complejas formadas por cadenas de aminoácidos.
La forma de la proteína está estrechamente relacionada con su función.
CASP is both the gold standard for assessing predictive techniques and a unique global community built on shared endeavour. Accuracy is measured on a range of 0-100 “GDT”. #AlphaFold has a median score of 92.4 GDT across all targets - its average error about the width of an atom. pic.twitter.com/cYCN12KxLZ
— DeepMind (@DeepMind) November 30, 2020
Impacto en el mundo
De acuerdo con el profesor Andrei Lupas, director del Instituto Max Planck de Biologías del Desarrollo y evaluador del CASP, las investigaciones de las estructuras de las proteínas podrían contribuir a nuestra comprensión de enfermedades específicas con un pequeño número de grupos de especialistas, por ejemplo: "ayudando a identificar proteínas que han fallado y razonando cómo interactúan".
"Estos conocimientos podrían permitir un trabajo más preciso en el desarrollo de fármacos, complementando los métodos experimentales existentes para encontrar tratamientos prometedores más rápido".
También está convencido de que la predicción podría ser útil en el esfuerzo por combatir la pandemia, ya que a principios del años predijeron varias estructuras proteicas del virus SARS-CoV-2.
El 'problema del plegamiento de proteínas'
En su discurso de aceptación del Premio Nobel de Química en 1972, Christian Anfinsen, postuló que en teoría la secuencia de aminoácidos de una proteína debería determinar completamente su estructura, hipótesis que provocó una búsqueda de cinco décadas para poder predecir gracias a los sistemas computacionales la estructura 3D de una proteína basándose en su secuencia de aminoácidos.
Podría interesarte: PODRÍAS PERDER TUS FOTOS DE GOOGLE PHOTO SI NO REALIZAS ESTA ACCIÓN